DNAの二本鎖は加熱やアルカリ条件で可逆的に解離します。この現象はPCRなどに利用されています。DNA の吸光度は濃度測定などに利用されます。また、DNA複製の校正機能、エキソンの選択性による翻訳産物の多様性、mRNA の成熟過程などは遺伝子発現・複製過程の精密さを実感できるメカニズムです。今回はこれらの現象について解説していきます。
Contents
DNA の物理・化学的性質
DNA 二本鎖の解離と会合
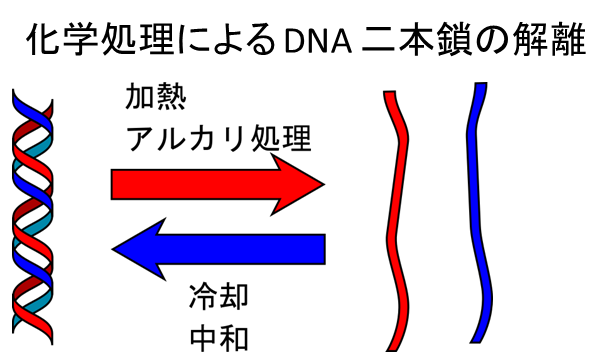
DNAの二本鎖は水素結合によって結びついています。加熱すると互いの分子運動が活発になって、水素結合を振り切ってしまうため二本鎖が解離します。この反応は可逆的であり、冷却することで元の二本鎖に戻ります。この現象は PCR などで利用されています。一方で、アルカリ性下に置くと水素結合が水酸基によって引っ張られて二本鎖を維持できなくなり、解離します。この現象も可逆的で、中和すれば元の二本鎖に戻ります。
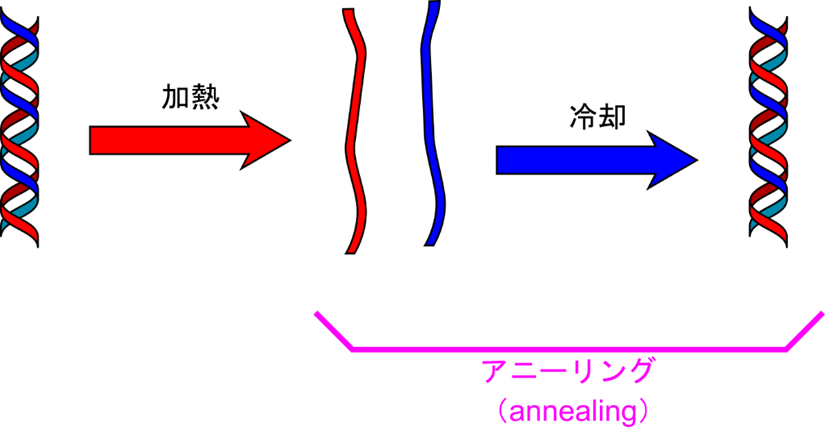
特に、DNA 溶液を加熱して二本鎖を解離させた場合、この溶液を冷却することで元の二本鎖に戻すことができます(図2)。この過程の内、冷却によって二本鎖を再会合させる過程のことをアニーリング(annealing)といいます。
DNAの吸光特性
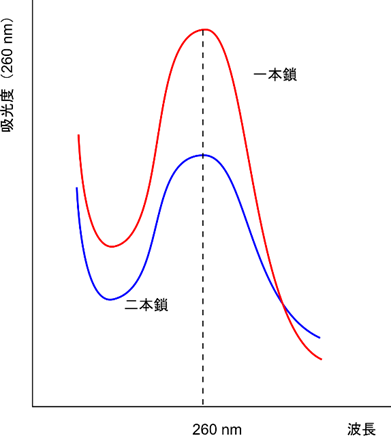
200 – 300 nm の範囲で DNA の吸光スペクトルを確認すると、260 nm にピークが現れます。このように、核酸は 260 nm に高い吸光度を示します(図3)。この特性は核酸の濃度を測定するために利用されています。
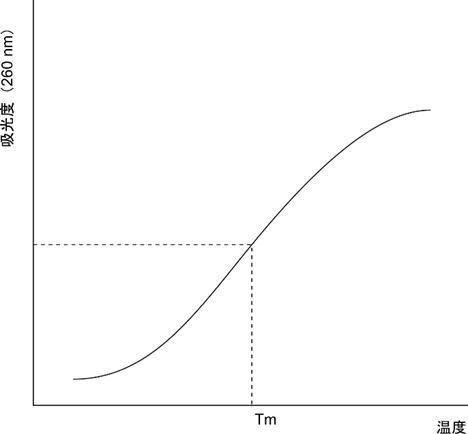
また、二本鎖の DNA よりも一本鎖の方が吸光度が高くなります。この特性を利用して、温度を上げながらDNA溶液の吸光度を測定すると、解離が進むにつれて吸光度が上昇します。解離が完了すると吸光度の上昇が止まり、この温度と吸光度の関係をグラフにするとシグモイド(S字)状になります。このとき、DNAの半分が解離する温度をTm値(melting temperature)と呼びます。Tm値はDNA断片の安定性やアニーリングの強さを示す指標として利用されます。
DNA の複製
DNA 複製メカニズム
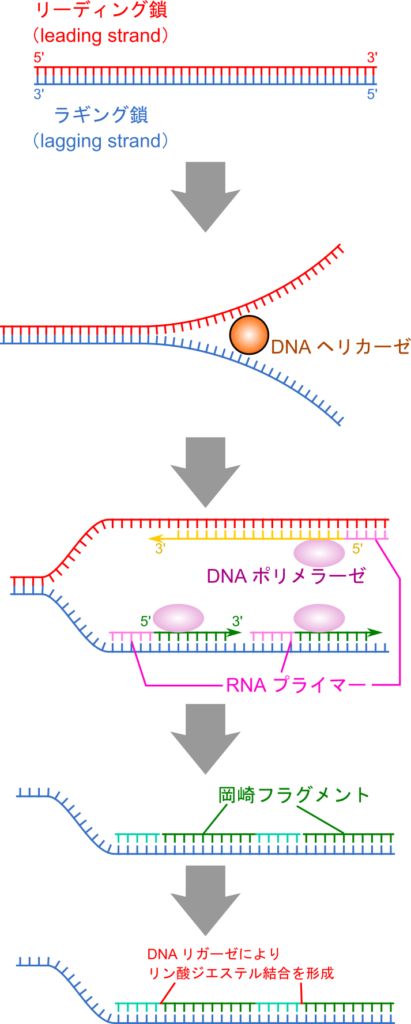
DNA の複製は、まずヘリカーゼが二本鎖をほどき、一本鎖になった DNA を鋳型として DNA ポリメラーゼが複製します。DNA の複製では、まずプライマーゼ(primase)が 短い RNA 断片(RNA プライマー;primer)を合成し、DNA ポリメラーゼが RNA プライマーを足掛かりにして伸長反応を行います。
リーディング鎖の伸長は連続的に進行しますが、ラギング鎖では断続的な複製が行われ、岡崎フラグメントと呼ばれる短いDNA断片が形成されます。最終的に、リガーゼがこれらの断片を繋ぎ合わせます。
DNA ポリメラーゼによる伸長反応は 5’→3′ 方向に行われるため、リーディング鎖の伸長反応は RNA プライマーから連続的に進みますが、ラギング鎖では複数個所に RNA プライマーが合成され、DNA ポリメラーゼが短いフラグメントごとに伸長反応を行って、岡崎フラグメント(okazaki fragment)と呼ばれる短いフラグメントを合成します。この伸長反応がRNA プライマーに到達すると、RNA プライマーを除去しながら DNA 鎖を合成し、最後に隣り合った岡崎フラグメント同士をリガーゼがつなぎ合わせます。
ポリメラーゼによる DNA の伸長反応は、リボースの 3′ 位の水酸基がリン酸基のリンを攻撃して、ピロリン酸が外れます。ピロリン酸はピロホスファターゼにより、無機リン酸二分子に分解されます。この反応は隣り合うリン酸基が切り離されているため、(ATP の分解同様)エネルギーを放出する反応です。
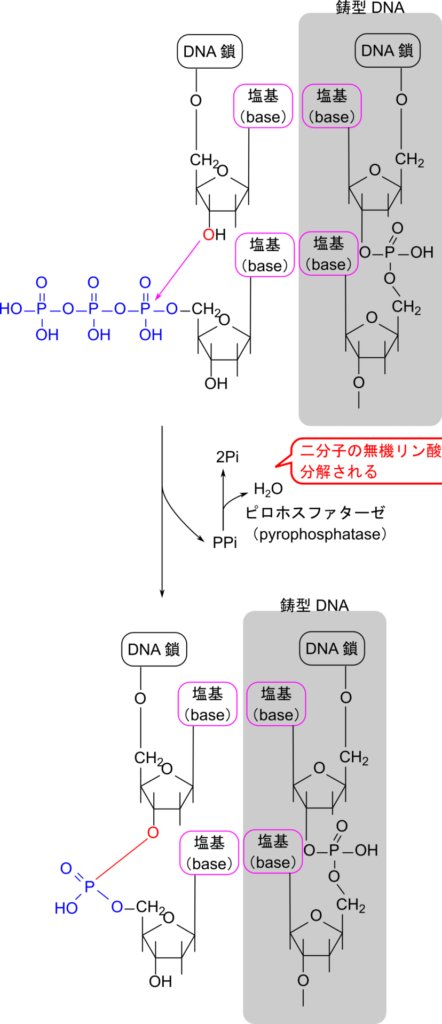
図6.DNA の伸長反応メカニズム
DNA の校正について
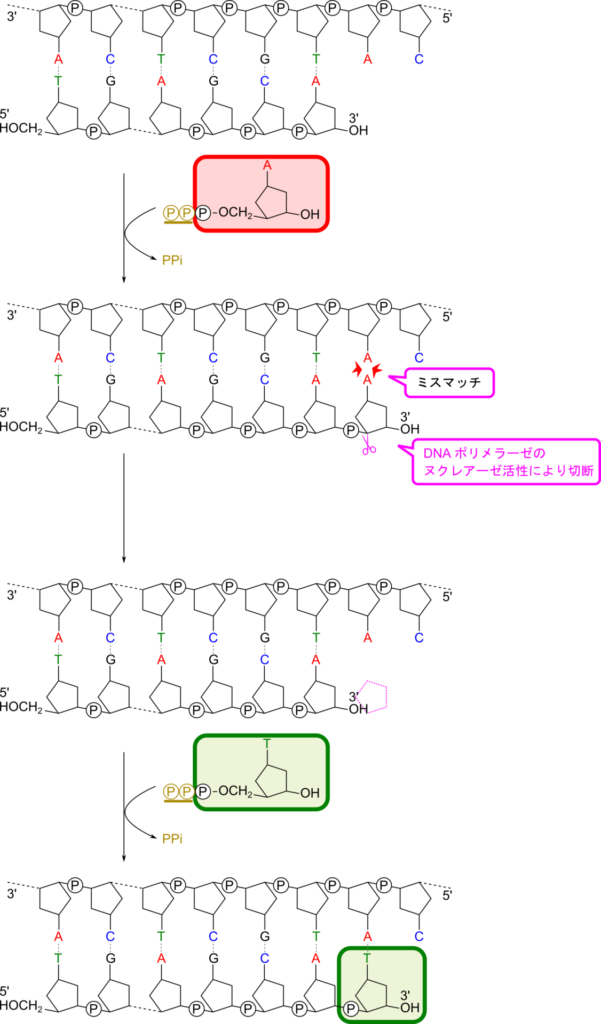
DNA の伸長で間違いが生じた場合は校正することが可能です。間違ったヌクレオチドが連結されてしまった場合、塩基対を形成できません。このようなヌクレオチドはポリメラーゼによる 3′-5′ ヌクレアーゼ活性(ヌクレオチドを切り離す活性)によって切除されます。その後、正しいヌクレオチドが連結されて校正が終了します。この校正機能により、DNAの複製は高い正確性を保っています。
遺伝子の発現
セントラルドグマ(central dogma)
遺伝子が発現する過程は、セントラルドグマとして知られています。まず、RNAポリメラーゼが遺伝子の配列をmRNAに転写(transcription)し、次にmRNAがリボソームで翻訳(translation)され、タンパク質が合成されます。
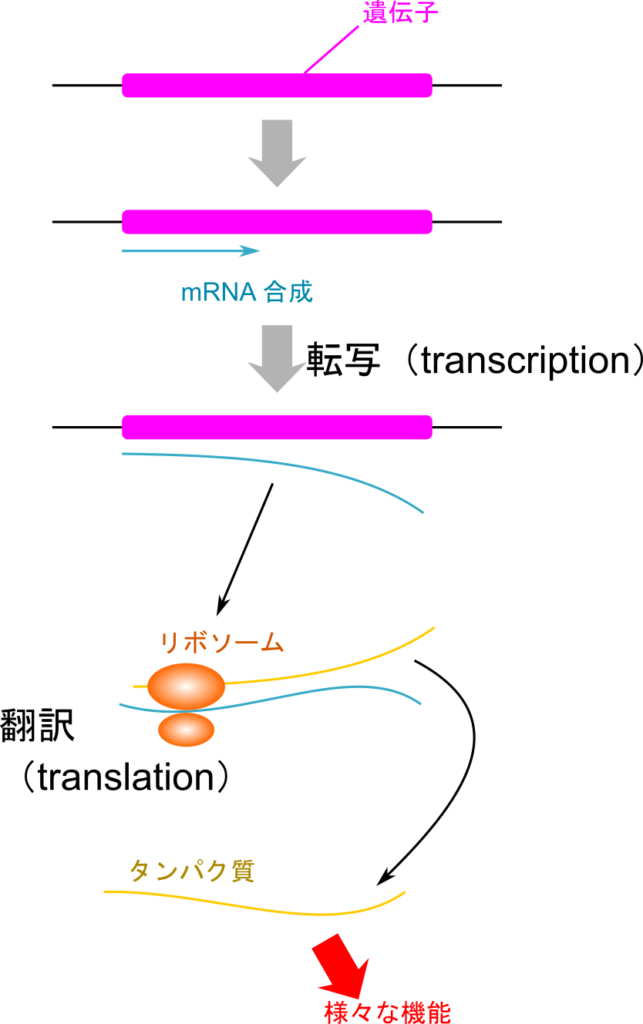
図8.遺伝子発現の過程
転写の制御
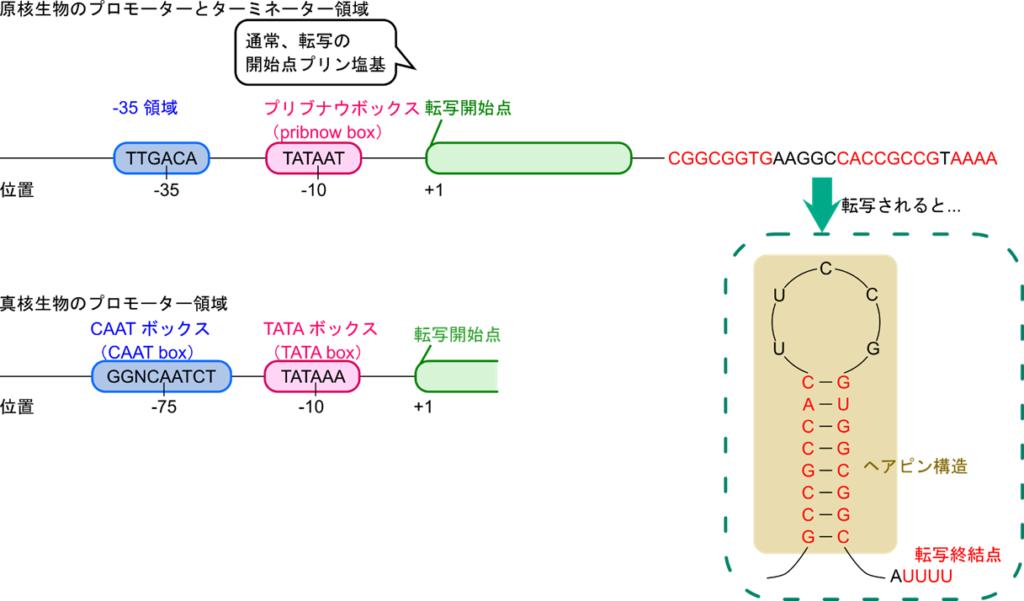
転写はプロモーターと呼ばれる遺伝子の上流領域にある特定の配列によって制御されます。プロモーターにはプリブナウボックス(-10塩基)やTATAボックスなどが存在し、RNAポリメラーゼが結合するポイントを提供します。
遺伝子発現はプロモーター(promoter)と呼ばれる遺伝子の上流領域にある特定の配列によって制御されます。この領域はRNA ポリメラーゼが結合する領域です。
原核生物のプロモーター配列
-10 塩基:TATAAT(プリブナウボックス;pribnow box)
-35 塩基:TTGACA
真核生物のプロモーター配列
-10 塩基:TATAAA
-75 塩基:GGNCAATCT
※- n 塩基は遺伝子の開始点を 1 とした場合の位置を示す。
-10 塩基は遺伝子の 10 塩基上流を意味する
mRNA の下流領域には GC が豊富で相補的な領域が存在します。この領域はターミネータ(terminator)と呼ばれています。mRNA はこの領域でヘアピン構造を形成し、RNA ポリメラーゼに転写終結点であることを認識させます。
このように、mRNA による転写は DNA 上の様々な配列によりその開始点や終結点がなど様々な指示を受けています。
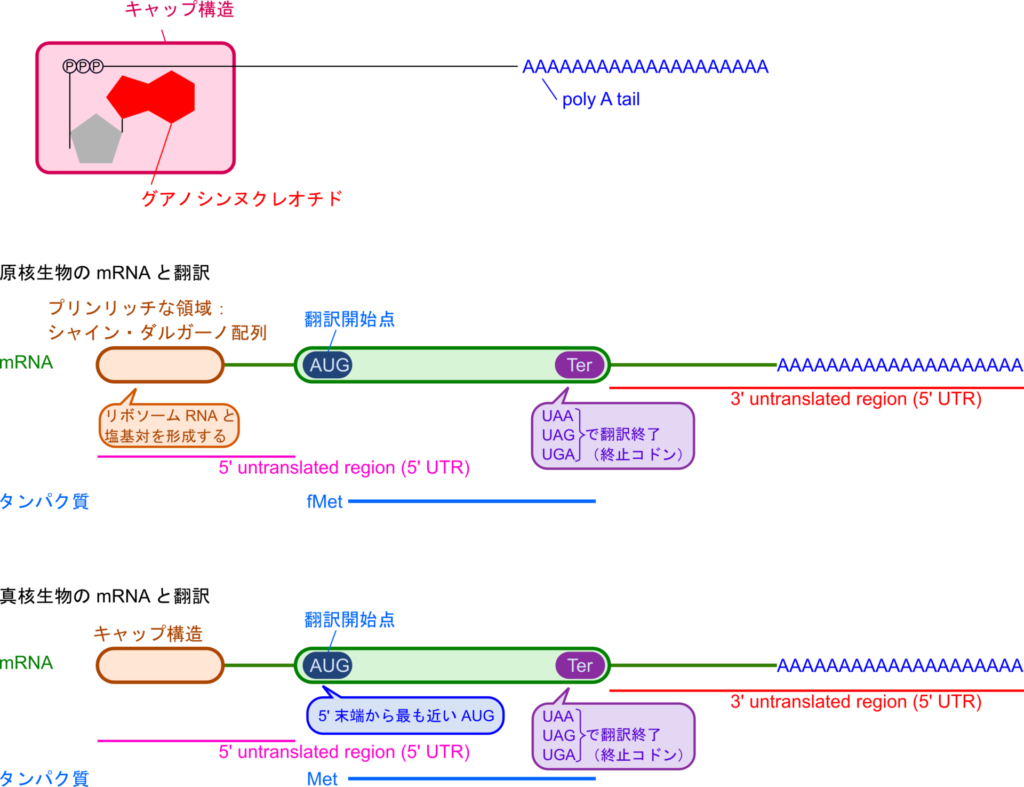
メッセンジャー RNA(mRNA)の構造と保護機能
mRNA は内部の情報を保護するため、5′ 末端、3′ 末端に特別な構造を有しています。mRNA の 5′ 末端はリン酸基3つを介してグアノシンヌクレオチドの 5′ 位が結合しています。この構造はキャップ構造と呼ばれています。一方で、 3′ 末端はアデニンが多数連続した領域があり、poly A tail と呼ばれています。mRNA はポリアデニル化シグナル(AATAAA)と GU の豊富な領域の間で切断され、ポリ(A)ポリメラーゼの作用により多数のアデニンが付加されて poly A tail が形成されます。
mRNA には非翻訳領域(untranslated region; UTR)と呼ばれる翻訳されない領域も含まれています。5’末端側の UTR を 5′ UTR、3′ 末端側の UTR を 3′ UTR といいます。翻訳領域は AUG で始まり、終止コドン(UAA、UAG、UGA)で終わります。翻訳領域では3塩基で一つのアミノ酸を指定しています。この3塩基のことをコドン(codon)といいます。
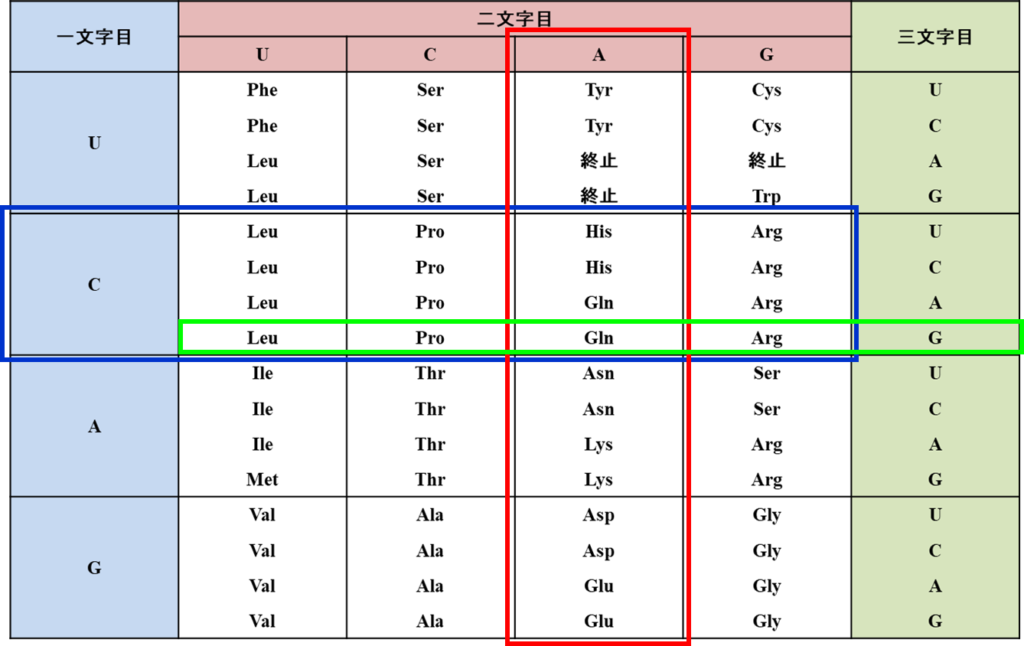
下に標準的なコドン表を記載します(表1)。「標準」というのはミトコンドリアでは異なるコドンが使用されているためです。
コドン CAG を例に読み方を見てみましょう。まず一文字目は C ですので、一文字目の列(青の列)を見て C の行(青枠)を見ます。二文字目は A ですので、二文字目の行(赤の行)を見て、A の列(赤枠)に注目します。最後に三文字目が G ですので、青枠と赤枠の交差する領域の三文字目が G に相当するアミノ酸残基を読み取り、Gln つまり、グルタミンが対応するとわかります。
ところで、塩基には 4 種類があります。塩基二つでとれる暗号のパターンは16通りあります。これでは20種類のアミノ酸をカバーできません。塩基三つでは64通りとなり、20種類を十分にカバーできます。さらに、異なるコドンが同一のアミノ酸をコードすること(コドンの縮重)により、突然変異によりコドンが書き換わったとしてもアミノ酸が変化してしまう可能性を低く抑えることができます。
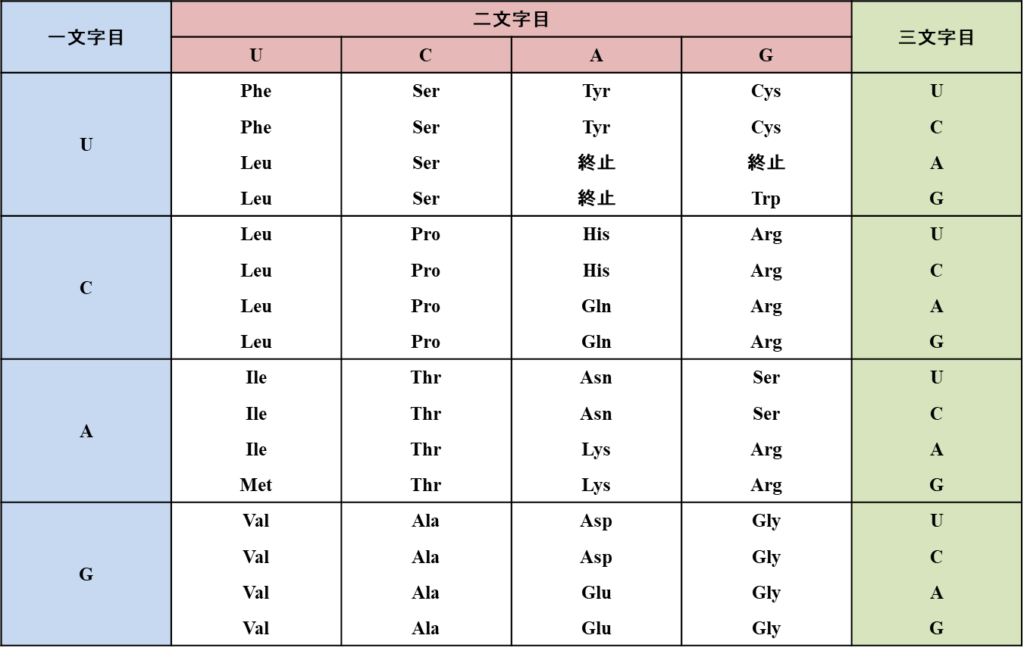
Ter: 終止コドン。三文字表記については以下の表のとおり。
| アミノ酸の三文字表記について | |
| Gly:グリシン(Glycine) | Thr:トレオニン(Threonine) |
| Ala:アラニン(Alanine) | Tyr:チロシン(Tyrosine) |
| Pro:プロリン(Proline) | Asn:アスパラギン(Asparagine) |
| Val:バリン(Valine) | Gln:グルタミン(Glutamine) |
| Leu:ロイシン(Leucine) | Cys:システイン(Cysteine) |
| Ile:イソロイシン(Isoleusine) | Lys:リシン(Lysine) |
| Met:メチオニン(Methionine) | Arg:アルギニン(Arginine) |
| Trp:トリプトファン(Tryptophan) | His:ヒスチジン(Histidine) |
| Phe:フェニルアラニン(Phenylalanine) | Asp:アスパラギン酸(Aspartic acid) |
| Ser:セリン(Serine) | Glu:グルタミン酸(Glutamic acid) |
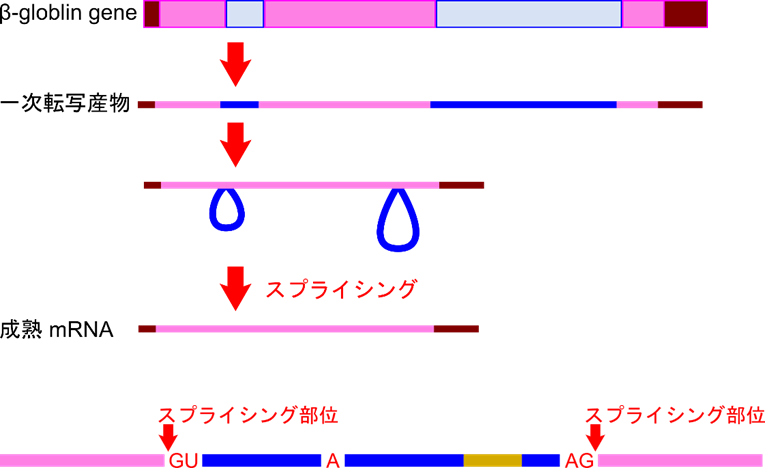
スプライシングによるイントロンの切除
一般に真核生物では遺伝子には翻訳される領域(エキソン;exon)と翻訳されない領域(イントロン;intron)が含まれています。遺伝子が転写されたのち、イントロン領域をスプライシングにより切除され、mRNA は成熟します。イントロンは GU で始まり、AG で終わります。この規則性は GT-AG 規則(GT-AG rule)といいます。また、GU-AG 間にアデニンを持っており、この構造がイントロンを切除するための認識領域になっています(図11)。
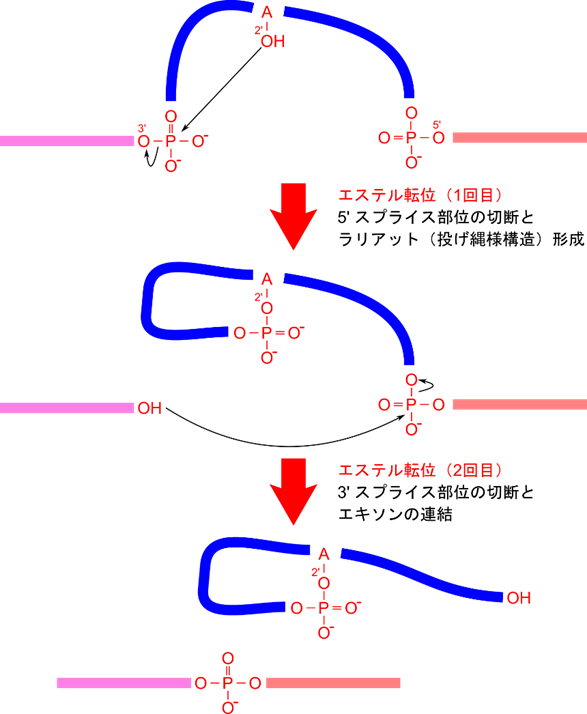
では、このようなスプライシングはどのようなメカニズムで行われているのでしょうか?イントロン内のアデニンの 2’位の水酸基がエキソンの 3′ 末端のリン酸基のリンを攻撃して結合を形成し、環状構造が形成されます。この構造は投げ輪のような恰好をしているのでラリアット(lariat )構造と呼ばれています。この後、切断された mRNA(の前半部分)の 3′ 末端の水酸基が次のエキソンの 5′ 末端のリン酸基のリンを攻撃して結合を形成し、イントロン部分が切除されます(図12)。
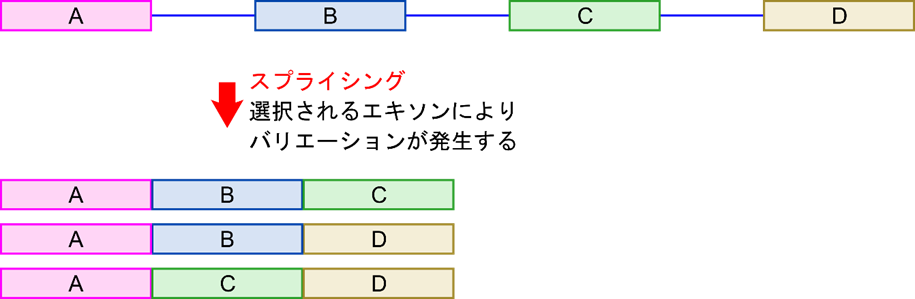
このイントロンの存在は一見すると無駄なように見えます。なぜ、イントロンが必要なのでしょうか?エキソンとイントロンの存在は、エキソンの選択によって翻訳産物のバリエーションを確保することに寄与しています。例えば、A、B、C、D のエキソンを持っている遺伝子について考えてみます。スプライシングの結果、A-B-C、A-B-D、A-C-D のエキソンが選択されると、一つの遺伝子から3つの翻訳産物ができたことになります。このように、イントロンのスプライシングの過程は同時にエキソンの選択の過程でもあるのです。この仕組みはオルタナティブスプライシング(alternative splicing)と呼ばれ、限られた遺伝子で多様な翻訳産物を生み出すことに寄与しています。スプライシングの結果得られたバリエーションはスプライシングバリアント(splicing variant)と呼ばれています。
今回の記事では、DNAの物理・化学的性質や複製、遺伝子発現のメカニズムについて解説しました。DNAの二本鎖の解離と再会合、吸光特性は核酸を利用した様々な試験系に利用される重要な特性です。そして後半に紹介した複製のプロセスは、生物学的な現象の根幹をなす重要な要素です。特に、複製の高精度を保つための校正機能や、スプライシングによる遺伝子の多様性創出は、生命の合理性を理解する上で欠かせない知識です。これらの基礎知見は今後、遺伝子工学や分子生物学などの分野に理解を進めていくうえでも重要な基礎知識となるでしょう。
参考文献
- 島原健三 (1991). 概説 生物化学. 三共出版. pp. 224-264
- Jeremy M. Berg, John L. Tymoczko, Gregory J. Gatto Jr., Lubert Stryer著、入村達郎、岡山博人、清水孝雄、中野徹訳 (2018). ストライヤー生化学 第8版. 東京化学同人. pp. 775-840
- T. A. Brown 著、西郷 薫 監訳(1999)分子遺伝学 第3版. 東京化学同人. pp. 64-85